転移RNA
| tRNA | |
|---|---|
| 識別 | |
| 略称 | t |
| Rfam | RF00005 |
| その他のデータ | |
| リボ核酸の種類 | gene, tRNA |
| PDB構造 | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow8, 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
転移RNA(てんいRNA、英: transfer RNA、tRNA)は[1]、通常76–90ヌクレオチド(真核生物の場合[2])のRNAからなるアダプター分子であり、遺伝情報を含むmRNAとタンパク質のアミノ酸配列とを物理的に結びつける役割を担う。運搬RNA、トランスファーRNAとも呼ばれ、通常tRNAと略記される。かつてはsRNA(soluble RNA)と呼ばれていた。tRNAは、細胞内のリボソームというタンパク質合成機械にアミノ酸を運ぶことでこれを行う。伝令RNA(メッセンジャーRNA、mRNA)上の3ヌクレオチドコドンと、tRNA上の3ヌクレオチドアンチコドンの相補的な関係が、mRNA上のコードに基づくタンパク質の合成に結びつく。このように、tRNAは、遺伝暗号に従って新しいタンパク質を生物学的に合成する翻訳に欠かせない要素である。
概要
[編集]
mRNA上に示されたヌクレオチド配列は、そのmRNAに転写された遺伝子のタンパク質産物にどのアミノ酸が組み込まれるかを特定する。これに対し、tRNAの役割は、遺伝暗号のどの配列がどのアミノ酸に対応するかを特定することである[3]。mRNAは、一連の連続したコドンとしてタンパク質をコード化し、それぞれのコドンは特定のtRNAによって識別される。tRNAの一端は、アンチコドンという3ヌクレオチド配列による遺伝暗号に一致する。このアンチコドンは、タンパク質生合成の際に、mRNA上のコドンと相補的な3塩基対を形成する。
tRNAのもう一方の端には、アンチコドン配列に対応するアミノ酸が共有結合している。個々の種類のtRNA分子は、それぞれ1種類のアミノ酸にしか結合できないため、各々の生物は多くの種類のtRNAを持っている。遺伝暗号には同じアミノ酸を指定する複数のコドンがあるため、同じアミノ酸を運ぶのにもかかわらず異なるアンチコドンを持つ異なるtRNA分子が存在する。
tRNAの3'末端への共有結合は、アミノアシルtRNA合成酵素と呼ばれる酵素によって触媒される。タンパク質合成の際、アミノ酸が結合したtRNAは、伸長因子と呼ばれるタンパク質によってリボソームに運ばれる。伸長因子は、tRNAとリボソームの結合、新しいポリペプチドの合成、およびmRNAに沿ったリボソームのトランスロケーション(転移)を助ける。tRNAのアンチコドンがmRNAと一致すると、すでにリボソームに結合している別のtRNAが、成長中のポリペプチド鎖を自身の3'末端から、新たに運ばれたtRNAの3'末端に結合したアミノ酸に転移させ、リボソームがこの反応を触媒する。tRNA分子内に含まれる多数のヌクレオチド(塩基)は化学修飾を受ける(修飾塩基)。メチル化や脱アミノなどがよく見られるほか、二重結合の還元やアミノ酸の付加、硫黄化など多くの修飾が存在する。それぞれの修飾塩基は、tRNAの適切な高次構造の安定化や、アミノアシルtRNA合成酵素の認識の変更、アンチコドンにおいて塩基対合の特性を変化させる役割などtRNAの機能を整える重要な働きをしている[4]。
構造
[編集]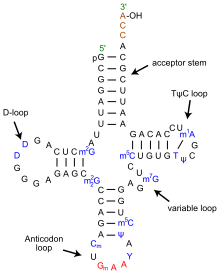

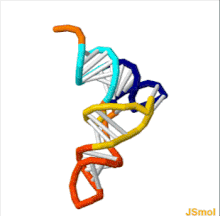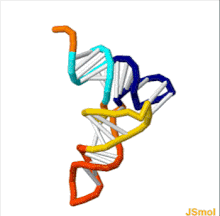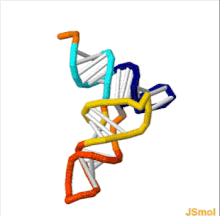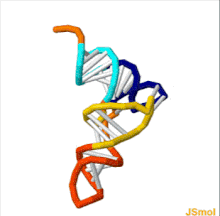
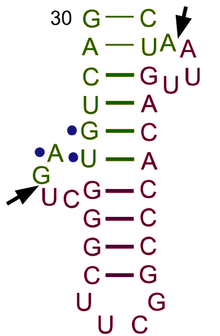
tRNAの構造は、一次構造、二次構造(通常クローバー葉構造(cloverleaf structure)として視覚化)、および三次構造(すべての種類のtRNAは、リボソームのP部位およびA部位に適合するように、同様のL字型の三次元構造を持っている)に分解することができる[6]。クローバー葉構造は、一般的なRNAの三次構造モチーフであるヘリックス(らせん)の同軸的スタッキングによって、L字型の三次元構造に折り畳まれる。 tRNA分子の各アーム(腕)の長さ、およびループの直径は、生物種によって異なる[6][7]。tRNAの構造は次のものからなる。
- 5′末端リン酸基
- 5′末端はリン酸基を持つ。
- アクセプターステム
- アクセプターステムは、L字型の短い側に相当し、5′末端ヌクレオチドと3′末端ヌクレオチド(アミノ酸を結合するために用いるCCA 3′末端基を含む)が塩基対を形成した7–9塩基対(bp)のステム(幹)である。一般に、このような3′末端tRNA様構造は「ゲノムタグ(genomic tags)」と呼ばれる。アクセプターステムは、非ワトソン=クリック型塩基対を含むことがある[6][8]。
- 3′末端CCA尾部
- CCA尾部は、tRNA分子の3′末端にあるシトシン-シトシン-アデニン配列のことである。アミノアシルtRNAを形成するためにアミノアシルtRNA合成酵素によってtRNAにロードされたアミノ酸は、CCA尾部の3′ヒドロキシル基と共有結合している[9]。この配列は、酵素によるtRNAの認識に重要であり、翻訳において重要な意味を持っている[10][11]。原核生物では、一部のtRNA配列にCCA配列が転写される。ほとんどの原核生物のtRNAや真核生物のtRNAでは、プロセシング中にCCA配列が付加されるため、tRNA遺伝子には現れない[12]。
- Dアーム
- Dアームは、4–6 bpのステムで、L字型の長い側の基部に相当する。しばしば修飾塩基としてジヒドロウリジンを含むループで終わる[6]。
- アンチコドンアーム
- アンチコドンアームは、5 bpのステムで、ループにアンチコドンを含む[6]。L字型の長い側の先端に相当する。tRNAの5'側から3'側への一次構造にはアンチコドンが含まれているが、mRNAを5′側から3′側へと読むためには3′側から5′側への方向性が必要なため、その順序が逆になっている。
- Tアーム
- Tアームは、4–5 bpのステムで、TΨCという配列を含むループで終わる。Ψはシュードウリジンで、修飾されたウリジンのことである[6]。L字型の関節部に相当する。
- 修飾塩基
- tRNA全体のいくつかの位置には、特にメチル化(例: tRNA(グアニン-N7)-)-メチルトランスフェラーゼによる)によって修飾された塩基が存在する。最初のアンチコドン塩基、すなわち「ゆらぎ位置(wobble-position)」は、イノシン(アデニン由来)、キューオシン(グアニン由来)、ウリジン-5-オキシ酢酸(ウラシル由来)、5-メチルアミノメチル-2-チオウリジン(ウラシル由来)、ライシジン(シトシン由来)に修飾されることがある[13]。
アンチコドン
[編集]アンチコドン(anticodon)は 3ヌクレオチドからなる単位であり、mRNAコドンの3塩基に対応する[14]。それぞれの種類のtRNAは、固有のアンチコドンの3連配列を持ち、アミノ酸に関する1つまたは複数のコドンに対して3つの相補的な塩基対を形成することができる。アンチコドンの中には、ゆらぎ塩基対によって複数のコドンと対をなすものもある。アンチコドンの最初のヌクレオチドは、mRNAには存在しないイノシンであることが多く、対応するコドン位置の複数の塩基と水素結合することができる[4]:29.3.9。遺伝暗号 (en:英語版) では一般的に、4種類の塩基と3つの位置によるすべての組み合わせ、あるいは少なくともピリミジン塩基とプリン塩基の両方によって、1つのアミノ酸が特定される。たとえば、アミノ酸のグリシンは、GGU、GGC、GGA、GGGというコドン配列でコードされている。また、ミトコンドリアのように、最初のアンチコドンの位置(ゆらぎ位置(wobble position)と呼ばれる)に別の修飾ヌクレオチドが現れて、遺伝暗号に微妙な変化を与えることもある[15]。標準遺伝暗号には61個のセンスコドンがあるので、tRNA分子をアミノ酸を指定するコドンと1対1で対応させるには、細胞あたり61種類のtRNAを持つ必要がある。しかし、ゆらぎ塩基は、特定のアミノ酸を指定するコドンのすべてというわけではないが、いくつかのコドンと結合することができるため、多くの細胞は61種類未満のtRNAしか持っていない。61個のセンスコドンをすべて明確に翻訳するためには、少なくとも31個のtRNAが必要である[3][16]。
アミノアシル化
[編集]アミノアシル化は、化合物にアミノアシル基を付加する過程であり、tRNA分子のCCA 3′末端にアミノ酸を共有結合させる。それぞれのtRNAは、アミノアシルtRNA合成酵素によって特定のアミノ酸が結合し、アミノアシル化される。1つのアミノ酸に対して複数のtRNAと複数のアンチコドンが存在する可能性があるという事実にもかかわらず、通常、各アミノ酸に対して1つのアミノアシルtRNA合成酵素が存在する。この合成酵素が適切なtRNAを認識するのは、アンチコドンのみを介して行われるだけではなく、アクセプターステムが重要な役割を果たすことが多い[17]。
反応:
ある種の生物では、1つまたは複数のアミノリン酸tRNA合成酵素が欠損していることがある。この結果、化学的に関連するアミノ酸がtRNAに結合するが、1つまたは複数の酵素を使用してtRNAが正しく結合するように修正される。たとえば、ヘリコバクター・ピロリでは、グルタミニルtRNA合成酵素が欠損している。そのため、グルタミン酸tRNA合成酵素が、tRNA-グルタミン(tRNAGln)にグルタミン酸を結合する。次に、アミド基トランスフェラーゼがグルタミン酸の酸側鎖をアミドに変換して、正しく結合したGln-tRNAGlnが形成される。
アミノアシル化の阻害は、ある種の疾患を治療する方法として有効となる可能性がある。たとえば、がん細胞は、健康な細胞と比較して、アミノアシル化の阻害に対して比較的脆弱な可能性がある。がんおよびウイルス生物学に関連するタンパク質合成は、特定のtRNA分子に大きく依存していることが多い。たとえば、肝臓がんの場合、tRNALysCUUにリジンを結合することで肝臓がん細胞の増殖や転移を維持しているが、健康な細胞では、細胞生理を維持するための、このtRNAに対する依存度ははるかに低い[18]。同様に、E型肝炎ウイルスは、感染していない細胞とは大きく異なるtRNA組成を必要とする[19]。したがって、特定のtRNA種のアミノアシル化を阻害することは、多くの疾患を合理的に治療する有望な新しい手段と考えられている。
リボソームへの結合
[編集]リボソームには、2つのリボソームサブユニット間にまたがるtRNA結合部位として、A部位(aminoacyl、アミノアシル)[21]、P部位(peptidyl、ペプチジル)、E部位(exit、出口)という3つの部位がある。さらにリボソームには、mRNAの解読時やタンパク質合成の開始時に使用されるtRNAの結合部位が2つあり、これらはT部位(伸長因子EF-Tuと命名)とI部位(initiation、開始)と呼ばれる[22][23]。慣例により、tRNA結合部位の表記は、リボソーム小サブユニット上の部位を先に記し、リボソーム大サブユニット上の部位を後に記す。たとえば、A部位はA/A、P部位はP/P、E部位はE/Eと表記されることが多い[22]。A部位やP部位におけるL27、L2、L14、L15、L16などの結合タンパク質は、A. P. Czernilofsky et al.によるアフィニティーラベリングによって決定されている(Proc. Natl. Acad. Sci, USA, pp. 230–234, 1974)。
翻訳開始が完了すると、最初のアミノアシルtRNAがP/P部位に位置し、次に述べる伸長サイクルの準備が整う。翻訳が伸長する過程では、まずtRNAが伸長因子Tu(EF-Tu)、もしくは真核生物や古細菌におけるその対応物(それぞれeEF-1、aEF-1)との複合体を形成してリボソームに結合する。この最初のtRNA結合部位をA/T部位と呼ぶ。A/T部位では、A部位と呼ぶ半分がmRNA解読部位があるリボソーム小サブユニットに存在する。mRNA解読部位は、翻訳の際にmRNAのコドンを読み出す場所である。T部位と呼ぶもう半分は主にリボソーム大サブユニットに存在し、EF-TuまたはeEF-1がリボソームと相互作用する。mRNAの解読が完了すると、アミノアシルtRNAはA/A部位に結合し、それに結合したアミノ酸に次のペプチド結合を形成する準備が整う[24]。A/A部位に結合したアミノアシルtRNAに成長するポリペプチドを転移させるペプチジルtRNAは、P/P部位に結合する。ペプチド結合が形成されると、P/P部位のtRNAはアシル化、すなわち3'末端が遊離し、A/A部位のtRNAは成長中のポリペプチド鎖を解離させる。次の伸長サイクルを可能にするため、tRNAは次にA/P・P/E混成の結合部位を移動し、サイクルを完了してP/P・E/E部位に位置するようになる。A/A・P/P部位のtRNAがP/P・E/E部位に移動すると、mRNAもコドン1つ分だけ移動し、A/T部位が空き、次のmRNA解読のための準備が整う。そして、E/E部位に結合したtRNAはリボソームから離脱する。
実際には、アミノアシルtRNAが最初に結合するのはP/I部位で、細菌ではIF2と呼ばれる開始因子によって送達される[23]。しかし、真核生物や古細菌のリボソームにおけるP/I部位の存在はまだ確認されていない。P部位におけるタンパク質L27は、E. Collatz and A. P. Czernilofskyによるアフィニティーラベリングによって決定されている(FEBS Lett., Vol. 63, pp. 283–286, 1976)。
tRNA遺伝子
[編集]ゲノムに含まれるtRNA遺伝子の数は、生物によってさまざまである。たとえば、遺伝学の研究で一般的なモデル生物である線虫カエノラブディティス・エレガンス(C. elegans)は、核ゲノムに29,647個の遺伝子を持ち[25]、そのうち620個がtRNAをコードしている[26][27]。出芽酵母(Saccharomyces cerevisiae)は、ゲノム中に275個のtRNA遺伝子を含む。
2013年1月の推計によると、ヒトゲノムではタンパク質コード遺伝子が合計で約20,848個あり[28]、細胞質tRNA分子をコードする核遺伝子が497個、tRNA由来の偽遺伝子(もはや機能しないと考えられているtRNA遺伝子[29])が324個ある(ただし、偽tRNAは細菌の抗生物質耐性に関与していることが示されている[30])。すべての真核生物と同様に、ヒトには22個のミトコンドリアtRNA遺伝子がある[31][要文献特定詳細情報]。これらの遺伝子の変異のいくつかは、MELAS症候群のような重篤な疾患と関連している。また、核染色体には、ミトコンドリアtRNA遺伝子と配列が非常に類似した領域が特定されている(tRNA-lookalikes、tRNA類似遺伝子)[32]。これらのtRNA類似遺伝子はまた、核ミトコンドリアDNA(ミトコンドリアから核に移された遺伝子)の一部とも考えられている[32][33]。こうした、ミトコンドリアtRNAの核内コピーが複数存在する現象は、ヒトからオポッサムまで多くの高等生物で観察されており[34]、類似遺伝子が機能している可能性を示唆している。
細胞質tRNA遺伝子は、アンチコドンの特徴によって49種類のファミリーに分類される。これらの遺伝子は、22番染色体とY染色体を除くすべての染色体上に見られる。また、1番染色体だけでなく、6番染色体にも大規模クラスター(140個のtRNA遺伝子)が観察されている[29]。
HGNCは、ゲノムtRNAデータベース(GtRNAdb)やこの分野の専門家と共同で、tRNAをコードするヒト遺伝子に対する固有の名称を承認した。
進化
[編集]tRNAの上半分(Tアームと、5'末端リン酸基と3'末端CCA基を持つアクセプターステムからなる)と下半分(Dアームと、アンチコドンアームからなる)は、構造的にも機能的に独立したユニットである。上半分は、初期のRNAワールドにおける複製用のtRNA様分子を標識付けていた可能性がある3′末端のゲノムタグを含めて、最初に進化したかもしれない。下半分は、たとえばRNAワールドでタンパク質合成として始まり、リボ核タンパク質ワールドに変わったように、後から拡張する形で進化したのかもしれない。これはゲノムタグ仮説(genomic tag hypothesis)として提案されたシナリオである。実際、tRNAやtRNA様凝集体は、現在でも複製における重要な触媒的な影響力(すなわちリボザイム)を果たしている。これらの役割は、RNAワールドの「分子化石(あるいは化学化石)」と見なすことができる[35]。
ゲノムtRNA含有量は、生命の生物学的ドメイン間でゲノムを区別する特徴である。古細菌は、ゲノムtRNA含有量が最も単純で、遺伝子のコピー数が均一であるのに対し、細菌は中間的で、真核生物は最も複雑な状況を示す[36]。真核生物は自然界における他の2つの生物界よりもtRNA遺伝子の含有量が多いだけでなく、異なるアイソアクセプター(isoacceptors)ごとに遺伝子コピー数が大きく変動しており、この複雑さは、tRNA遺伝子の重複やアンチコドン特異性の変化によるものではないかと考えられる[要出典]。
異なる生物種間におけるtRNA遺伝子コピー数の進化は、所定のtRNAの解読能力を向上させる特定のtRNA修飾酵素(細菌ではウリジンメチルトランスフェラーゼ、真核生物ではアデノシンデアミナーゼ)の出現と関連している[36]。たとえば、tRNAAlaは4種類のtRNAアイソアクセプター(AGC、UGC、GGC、CGC)をコード化する。真核生物では、AGCアイソアクセプターの遺伝子コピー数が他のアイソアクセプターと比べて非常に多く、これは、そのゆらぎ塩基がAからIへ修飾されていることと相関している。これと同じ傾向が、真核生物種のほとんどのアミノ酸で示されている。実際、これら2つのtRNA修飾の効果は、コドン使用量の偏りにも現れている。高発現遺伝子は、これらの修飾tRNAによって解読されるコドンのみを使用するコドンに富んでいるようであり、このことは、これらのコドンさらにはこれらのtRNA修飾が翻訳効率に関与している可能性を示唆している[36]。
多くの生物種が進化の過程で特定のtRNAを失っていることに留意するのは重要である。たとえば、哺乳類と鳥類は64種類あるtRNA遺伝子のうち14種類を欠いているが、他の生物はこれらのtRNAを保有している[37]。tRNAが完全に一致しないコドンを翻訳するために、生物はウォブリング(wobbling)と呼ばれる戦略に頼る。ウォブリングは、tRNA/mRNAの組が不完全な一致でも翻訳を行わせるが、この戦略は翻訳エラーの傾向を増やす[38]。進化の過程でtRNA遺伝子が失われた理由については議論が続いているが、ウイルス感染に対する抵抗性の向上と関係している可能性がある[39]。ヌクレオチド3塩基配列は、アミノ酸と関連するtRNAの種類よりも多くの組み合わせを提示できるため、遺伝暗号には冗長性があり、同じアミノ酸を複数の異なる3塩基コドンで表現することができる。このコドンの偏りは、コドン最適化を必要とするもので、標的生物のtRNA目録のコード配列を調べることで、発現系に応じたコドン交換が可能になる[40]。
tRNA由来フラグメント
[編集]tRNA由来フラグメント(tRNA-derived fragments、tRF)は、成熟tRNAまたは前駆体転写産物が切断された後に出現する短い分子である[41][42][43][44]。細胞質tRNAもミトコンドリアtRNAもフラグメントを生成することができる[45]。成熟したtRNAに由来すると考えられているtRFには、比較的長いtRNAの半分と、短い5'-tRF、3'-tRF、およびi-tRFの少なくとも4種類の構造型が存在する[41][45][46]。前駆体tRNAは切断され、5'リーダー配列や3'トレイル配列から分子を生成することができる。切断酵素としては、アンジオゲニン、Dicer、RNase Z、およびRNase Pがあげられる[41][42]。特にアンジオゲニンの場合、tRFは、その3'末端に環状リン酸、5'末端にヒドロキシル基という特徴的な構造を持っている[47]。tRFは、RNA干渉、特にtRNAをプライマーとして複製を行うレトロウイルスやレトロトランスポゾンの抑制に関与しているように思われる。アンジオゲニンによって切断されたハーフtRNAは、tiRNAともいう。piRNAとして機能するものも含む、より小さな断片の生合成については、あまり理解されていない[48]。
tRFは、複数の依存性と役割をもっており、性別間、人種間、あるいは疾病状態間で大きな変化を示す[45][49][50]。機能的には、AgoにロードされRNAi経路を介した作用や[43][46][51]、ストレス顆粒の形成への関与や[52]、RNA結合タンパク質からmRNAを置換する[53]、翻訳の阻害などがあげられる[54]。システムあるいは生体レベルでは、4種類のtRFは多様な活動範囲を持っている。機能的には、tRFはウイルス感染[55]、癌(がん)[46]、細胞増殖に関連し[47]、またエピジェネティックな世代を超えた代謝調節にも関与している[56]。
tRFはヒトに限定されず、さまざまな生物に存在することが確認されている[46][57][58][59]。
tRFについてより詳しく知りたい場合、2つのオンラインツールを利用できる。ミトコンドリアおよび核内tRNAフラグメントの対話的に探索するためのフレームワーク (MINTbase) と[60][61]、転移RNA関連フラグメントのリレーショナルデータベース (tRFdb) である[62]。また、MINTbaseはゲノムに依存しないtRFライセンスプレート (またはMINTcodes)と呼ばれるtRFの命名スキームを提供しており、これはRNA配列をより短い文字列に圧縮するものである。
合成tRNA
[編集]合成サプレッサー伸長tRNAは、遺伝子のコード配列に配置されたナンセンスコドンに非天然アミノ酸を組み込むために用いられる。アンバー終止コドンUAGで翻訳を開始するために、合成開始tRNA(metY遺伝子がコードするCUAアンチコドンを持つtRNAfMet2)が用いられている。この種の合成tRNA(engineered tRNA)は、UAGコドンで通常起こる翻訳停止シグナルを抑制することから、ナンセンスサプレッサーtRNAと呼ばれている。アンバー開始tRNAは、強力なシャイン・ダルガノ配列が先行するUAGコドンに、メチオニン[63]とグルタミン[64]を挿入する。このアンバー開始tRNAの研究により、この系は通常のAUG開始コドンと直交しており、ゲノム再コード化された大腸菌株でオフターゲット翻訳開始イベントが検出されないことが示された[63]。
tRNAの生合成
[編集]真核細胞では、tRNAは核内でRNAポリメラーゼIIIによってpre-tRNAとして転写される[65]。RNAポリメラーゼIIIは、tRNA遺伝子内の5′遺伝子内調節領域(5′-ICR、D調節領域、Aボックス)と3′-ICR(T調節領域、Bボックス)の、2つの高度に保存された下流プロモーター配列を認識する[2][66][67]。第1プロモーターは成熟tRNAの+8から始まり、第2プロモーターは第1プロモーターの30–60ヌクレオチド下流に位置する。転写は、4個以上のチミジンの連続区間の後に終了する[2][67]。
pre-tRNAは、核内で広範な修飾を受ける。pre-tRNAの中には、機能的なtRNA分子を形成するためにスプライシングまたは切断されるイントロンを含むものがある[68]。細菌ではこれらを自己スプライシングするが、真核生物や古細菌ではtRNAスプライシングエンドヌクレアーゼによって除去される[69]。真核生物のpre-tRNAには、エンドヌクレアーゼによるtRNAイントロンの認識と正確なスプライシングをするのに重要なバルジ-ヘリックス-バルジ(BHB)構造モチーフが存在する[70]。このモチーフの位置と構造は進化的に保存されている。しかし、単細胞藻類など一部の生物には、BHBモチーフの位置やスプライシングされたイントロン配列の5′末端や3′末端が非標準であるものもある[70]。5′配列はRNase Pによって除去され[71]、3′末端はtRNase Z酵素によって除去される[72]。顕著な例外が古細菌のナノアルカエウム・エクウィタンス(Nanoarchaeum equitans)で見られ、RNase P酵素を持たず、成熟tRNAの5′末端から転写が始まるようにプロモーターが配置されている[73]。ヌクレオチジルトランスフェラーゼによって、テンプレート化されていない3′CCA尾部が付加される[74]。tRNAがLos1/Xpo-tによって細胞質へ輸送される前に[75][76]、tRNAはアミノアシル化される[77]。プロセシング過程の順序は保存されていない。たとえば、酵母では、スプライシングは核内ではなく、ミトコンドリア膜の細胞質側で行われる[78]。
それにもかかわらず、2021年3月、研究者らは、転移RNAの予備的な形態が、生命のごく初期の発生、すなわち生命起源における複製分子であった可能性を示唆する証拠を報告した[79][80]。
歴史
[編集]tRNAの存在は、RNAアルファベットからタンパク質アルファベットへの翻訳を媒介するアダプター分子が存在するはずだという仮定に基づいて、フランシス・クリックにより「アダプター仮説(adaptor hypothesis)」として最初に提唱された。ポール・C・ザメクニックとマーロン・ホーグランドがtRNAを発見した[81]。1960年代初頭、ボストンの2人の研究者、アレクサンダー・リッチとドナルド・キャスパー、プリンストン大学のジャック・R・フレスコのグループ、キングス・カレッジ・ロンドンの英国グループによって、構造に関する重要な研究が行われた[82]。1965年、コーネル大学のロバート・W・ホリーが一次構造を報告し、3つの二次構造が示唆された[83]。ウィスコンシン州のマディソンで、ロバート・M・ボックによって初めてtRNAが結晶化された[84]。その後の数年間に、いくつかの研究によってクローバー葉構造が確認され[85]、1974年にX線結晶構造解析を用いて最終的に確認された。2つの独立したグループ、アレクサンダー・リッチ率いるキム・ソンホウと、アーロン・クルーグ率いる英国のグループが、1年以内に同じ結晶学的な発見を発表した[86][87]。
訳語について
[編集]学術用語集では植物学編・遺伝学編が「転移RNA(運搬RNA)」、動物学編が「運搬RNA」としている(すべて増訂版)。JISの生体工学用語(K3610)では「転移RNA」である。一般には「転移RNA」の方が好んで用いられる傾向にあるが、高校教育では「運搬RNA」が用いられている。
参照項目
[編集]- 翻訳 (生物学) - 細胞で遺伝情報がmRNAに転写されてタンパク質を合成する過程
- 転写 (生物学) - DNAの断片をRNAにコピーする過程
- リボソーム - 細胞内で生体タンパク質の合成(mRNAの翻訳)を行う高分子
- アミノアシルtRNA合成酵素
- タンパク質生合成
- ノンコーディングRNA (ncRNA) - タンパク質に翻訳されないRNA分子
- イントロン - 転写はされるが最終的なRNA産物からは除かれる遺伝子内の塩基配列
- 修飾塩基 - 転写後修飾を受けたヌクレオチド
- ゆらぎ仮説 - 必要なコドンの種類に対してtRNAの種類がより少ないという事実を説明するための仮説
- tRNAのクローバー葉モデル
- 転移RNA様構造 - tRNAと類似する三次構造を持つRNA配列
- 滑りやすい配列 - リボソームのフレームシフトの速度と可能性を制御するコドンヌクレオチド配列の小領域
- mRNA - 遺伝子の塩基配列に対応する一本鎖のRNA分子
- アミノアシルtRNA - 同族アミノ酸が化学的に結合しているtRNA
- tmRNA - tRNAとmRNAの性質の両方を持つ細菌由来のRNA分子
脚注
[編集]- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (October 1965). “Production of antibodies to soluble RNA (sRNA)”. Proceedings of the National Academy of Sciences of the United States of America 54 (4): 1281–1285. Bibcode: 1965PNAS...54.1281P. doi:10.1073/pnas.54.4.1281. PMC 219862. PMID 5219832.
- ^ a b c Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). “Structure and transcription of eukaryotic tRNA genes”. CRC Critical Reviews in Biochemistry 19 (2): 107–144. doi:10.3109/10409238509082541. PMID 3905254.
- ^ a b Crick FH (December 1968). “The origin of the genetic code”. Journal of Molecular Biology 38 (3): 367–379. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ a b Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W. H. Freeman. ISBN 978-0-7167-4955-4
- ^ “Transfer RNA (tRNA)”. Proteopedia.org. 7 November 2018閲覧。
- ^ a b c d e f Itoh Y, Sekine S, Suetsugu S, Yokoyama S (July 2013). “Tertiary structure of bacterial serenocysteine tRNA”. Nucleic Acids Research 41 (13): 6729–6738. doi:10.1093/nar/gkt321. PMC 3711452. PMID 23649835.
- ^ Goodenbour JM, Pan T (29 October 2006). “Diversity of tRNA genes in eukaryotes”. Nucleic Acids Research 34 (21): 6137–6146. doi:10.1093/nar/gkl725. PMC 1693877. PMID 17088292.
- ^ Jahn M, Rogers MJ, Söll D (July 1991). “Anticodon and acceptor stem nucleotides in tRNA(Gln) are major recognition elements for E. coli glutaminyl-tRNA synthetase”. Nature 352 (6332): 258–260. Bibcode: 1991Natur.352..258J. doi:10.1038/352258a0. PMID 1857423.
- ^ Ibba M, Soll D (June 2000). “Aminoacyl-tRNA synthesis”. Annual Review of Biochemistry 69 (1): 617–650. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ^ Sprinzl M, Cramer F (1979). “The -C-C-A end of tRNA and its role in protein biosynthesis”. Progress in Nucleic Acid Research and Molecular Biology 22: 1–69. doi:10.1016/s0079-6603(08)60798-9. ISBN 978-0-12-540022-0. PMID 392600.
- ^ Green R, Noller HF (1997). “Ribosomes and translation”. Annual Review of Biochemistry 66: 679–716. doi:10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J (September 1990). “Isolation of a temperature-sensitive mutant with an altered tRNA nucleotidyltransferase and cloning of the gene encoding tRNA nucleotidyltransferase in the yeast Saccharomyces cerevisiae”. The Journal of Biological Chemistry 265 (27): 16216–16220. doi:10.1016/S0021-9258(17)46210-7. PMID 2204621.
- ^ McCloskey JA, Nishimura S (November 1977). “Modified nucleosides in transfer RNA”. Accounts of Chemical Research 10 (11): 403–410. doi:10.1021/ar50119a004.
- ^ Felsenfeld G, Cantoni GL (May 1964). “Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA”. Proceedings of the National Academy of Sciences of the United States of America 51 (5): 818–826. Bibcode: 1964PNAS...51..818F. doi:10.1073/pnas.51.5.818. PMC 300168. PMID 14172997.
- ^ Suzuki T, Suzuki T (June 2014). “A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs”. Nucleic Acids Research 42 (11): 7346–7357. doi:10.1093/nar/gku390. PMC 4066797. PMID 24831542.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Biology of the Cell. WH Freeman: New York. 5th ed.[要ISBN][要ページ番号]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (October 1993). “An operational RNA code for amino acids and possible relationship to genetic code”. Proceedings of the National Academy of Sciences of the United States of America 90 (19): 8763–8768. Bibcode: 1993PNAS...90.8763S. doi:10.1073/pnas.90.19.8763. PMC 47440. PMID 7692438.
- ^ Zhang R, Noordam L, Ou X, Ma B, Li Y, Das P, Shi S, Liu J, Wang L, Li P, Verstegen MM, Reddy DS, van der Laan LJ, Peppelenbosch MP, Kwekkeboom J, Smits R, Pan Q (January 2021). “The biological process of lysine-tRNA charging is therapeutically targetable in liver cancer”. Liver Int. 41 (1): 206–219. doi:10.1111/liv.14692. PMC 7820958. PMID 33084231.
- ^ Ou X, Ma B, Zhang R, Miao Z, Cheng A, Peppelenbosch MP, Pan Q (June 2020). “A simplified qPCR method revealing tRNAome remodeling upon infection by genotype 3 hepatitis E virus”. FEBS Letters 594 (12): 2005–2015. doi:10.1002/1873-3468.13764. PMID 32133647.
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (May 2011). “Structures of the bacterial ribosome in classical and hybrid states of tRNA binding”. Science 332 (6032): 981–984. Bibcode: 2011Sci...332..981D. doi:10.1126/science.1202692. PMC 3176341. PMID 21596992.
- ^ Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (January 2004). “Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and Mg2+-dependent interactions”. RNA 10 (1): 90–101. doi:10.1261/rna.5142404. PMC 1370521. PMID 14681588.
- ^ a b Agirrezabala X, Frank J (August 2009). “Elongation in translation as a dynamic interaction among the ribosome, tRNA, and elongation factors EF-G and EF-Tu”. Quarterly Reviews of Biophysics 42 (3): 159–200. doi:10.1017/S0033583509990060. PMC 2832932. PMID 20025795.
- ^ a b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (June 2005). “The cryo-EM structure of a translation initiation complex from Escherichia coli”. Cell 121 (5): 703–712. doi:10.1016/j.cell.2005.03.023. PMID 15935757.
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). “The Peptidyl Transferase Center: a Window to the Past”. Microbiol Mol Biol Rev 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ WormBase web site, http://www.wormbase.org Archived 2017-04-20 at the Wayback Machine., release WS187, date 25-Jan-2008.
- ^ Spieth J, Lawson D (January 2006). “Overview of gene structure”. WormBook: 1–10. doi:10.1895/wormbook.1.65.1. PMC 4781370. PMID 18023127.
- ^ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Genetics: From Genes to Genomes 2nd ed. McGraw-Hill: New York. p. 264.
- ^ Ensembl release 70 - Jan 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core Archived 2013-12-15 at the Wayback Machine.
- ^ a b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, etal (February 2001). “Initial sequencing and analysis of the human genome”. Nature 409 (6822): 860–921. Bibcode: 2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). “A pseudo-tRNA modulates antibiotic resistance in Bacillus cereus”. PLOS ONE 7 (7): e41248. Bibcode: 2012PLoSO...741248R. doi:10.1371/journal.pone.0041248. PMC 3399842. PMID 22815980.
- ^ Ibid. p. 529.
- ^ a b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). “Nuclear and mitochondrial tRNA-lookalikes in the human genome”. Frontiers in Genetics 5: 344. doi:10.3389/fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C (November 2011). “Nuclear insertions of mitochondrial origin: Database updating and usefulness in cancer studies”. Mitochondrion 11 (6): 946–953. doi:10.1016/j.mito.2011.08.009. PMID 21907832.
- ^ Telonis AG, Kirino Y, Rigoutsos I (2015). “Mitochondrial tRNA-lookalikes in nuclear chromosomes: Could they be functional?”. RNA Biol 12 (4): 375–380. doi:10.1080/15476286.2015.1017239. PMC 4615777. PMID 25849196.
- ^ Nancy Maizels and Alan M. Weiner: The Genomic Tag Hypothesis – What Molecular Fossils Tell Us about the Evolution of tRNA, in: The RNA World, Second Edition. 1999 Cold Spring Harbor Laboratory Press ISBN 978-0-87969-561-3/99, PDF
- ^ a b c Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (March 2012). “A role for tRNA modifications in genome structure and codon usage”. Cell 149 (1): 202–213. doi:10.1016/j.cell.2012.01.050. PMID 22464330.
- ^ Ou X, Peng W, Yang Z, Cao J, Wang M, Peppelenbosch MP, Pan Q, Cheng A (November 2020). “Evolutionarily missing and conserved tRNA genes in human and avian.”. Infect. Genet. Evol. 85: 104460. doi:10.1016/j.meegid.2020.104460. PMID 32679345.
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (March 2019). “Errors in translational decoding: tRNA wobbling or misincorporation?”. PLOS Genetics 15 (3): 2979–2986. doi:10.1371/journal.pgen.1008017. PMC 3158919. PMID 21930591.
- ^ Ou X, Wang M, Mao S, Cao J, Cheng A, Zhu D, Chen S, Jia R, Liu M, Yang Q, Wu Y, Zhao X, Zhang S, Liu Y, Yu Y, Zhang L, Chen X, Peppelenbosch MP, Pan Q (July 2018). “Incompatible Translation Drives a Convergent Evolution and Viral Attenuation During the Development of Live Attenuated Vaccine”. Front. Cell. Infect. Microbiol. 8: 249. doi:10.3389/fcimb.2018.00249. PMC 6058041. PMID 30073153.
- ^ van Warmerdam, T.. “YourBioHelper.com”. YourBioHelper. 2022年9月25日閲覧。
- ^ a b c Gebetsberger J, Polacek N (December 2013). “Slicing tRNAs to boost functional ncRNA diversity”. RNA Biology 10 (12): 1798–1806. doi:10.4161/rna.27177. PMC 3917982. PMID 24351723.
- ^ a b Shigematsu M, Honda S, Kirino Y (2014). “Transfer RNA as a source of small functional RNA”. Journal of Molecular Biology and Molecular Imaging 1 (2): 8. PMC 4572697. PMID 26389128.
- ^ a b Sobala A, Hutvagner G (2011). “Transfer RNA-derived fragments: origins, processing, and functions”. Wiley Interdisciplinary Reviews: RNA 2 (6): 853–862. doi:10.1002/wrna.96. hdl:10453/18187. PMID 21976287.
- ^ Keam SP, Hutvagner G (November 2015). “tRNA-Derived Fragments (tRFs): Emerging New Roles for an Ancient RNA in the Regulation of Gene Expression”. Life 5 (4): 1638–1651. doi:10.3390/life5041638. PMC 4695841. PMID 26703738.
- ^ a b c Telonis AG, Loher P, Honda S, Jing Y, Palazzo J, Kirino Y, Rigoutsos I (July 2015). “Dissecting tRNA-derived fragment complexities using personalized transcriptomes reveals novel fragment classes and unexpected dependencies”. Oncotarget 6 (28): 24797–822. doi:10.18632/oncotarget.4695. PMC 4694795. PMID 26325506.
- ^ a b c d Kumar P, Anaya J, Mudunuri SB, Dutta A (October 2014). “Meta-analysis of tRNA derived RNA fragments reveals that they are evolutionarily conserved and associate with AGO proteins to recognize specific RNA targets”. BMC Biology 12: 78. doi:10.1186/s12915-014-0078-0. PMC 4203973. PMID 25270025.
- ^ a b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (July 2015). “Sex hormone-dependent tRNA halves enhance cell proliferation in breast and prostate cancers”. Proceedings of the National Academy of Sciences of the United States of America 112 (29): E3816–E3825. Bibcode: 2015PNAS..112E3816H. doi:10.1073/pnas.1510077112. PMC 4517238. PMID 26124144.
- ^ Schorn, AJ; Martienssen, R (October 2018). “Tie-Break: Host and Retrotransposons Play tRNA.”. Trends in Cell Biology 28 (10): 793–806. doi:10.1016/j.tcb.2018.05.006. PMC 6520983. PMID 29934075.
- ^ Telonis AG, Rigoutsos I (March 2018). “Race Disparities in the Contribution of miRNA Isoforms and tRNA-Derived Fragments to Triple-Negative Breast Cancer”. Cancer Res 78 (5): 1140–54. doi:10.1158/0008-5472.CAN-17-1947. PMC 5935570. PMID 29229607.
- ^ Telonis AG, Loher P, Magee R, Pliatsika V, Londin E, Kirino Y, Rigoutsos I (Jun 2019). “tRNA Fragments Show Intertwining with mRNAs of Specific Repeat Content and Have Links to Disparities”. Cancer Res 79 (12): 3034–49. doi:10.1158/0008-5472.CAN-19-0789. PMC 6571059. PMID 30996049.
- ^ Shigematsu M, Kirino Y (2015). “tRNA-Derived Short Non-coding RNA as Interacting Partners of Argonaute Proteins”. Gene Regulation and Systems Biology 9: 27–33. doi:10.4137/GRSB.S29411. PMC 4567038. PMID 26401098.
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (April 2010). “Angiogenin-induced tRNA-derived stress-induced RNAs promote stress-induced stress granule assembly”. The Journal of Biological Chemistry 285 (14): 10959–10968. doi:10.1074/jbc.M109.077560. PMC 2856301. PMID 20129916.
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (May 2015). “Endogenous tRNA-Derived Fragments Suppress Breast Cancer Progression via YBX1 Displacement”. Cell 161 (4): 790–802. doi:10.1016/j.cell.2015.02.053. PMC 4457382. PMID 25957686.
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (August 2011). “Angiogenin-induced tRNA fragments inhibit translation initiation”. Molecular Cell 43 (4): 613–623. doi:10.1016/j.molcel.2011.06.022. PMC 3160621. PMID 21855800.
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (January 2015). “Small tRNA-derived RNAs are increased and more abundant than microRNAs in chronic hepatitis B and C”. Scientific Reports 5: 7675. Bibcode: 2015NatSR...5E7675S. doi:10.1038/srep07675. PMC 4286764. PMID 25567797.
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R, Mello CC, Garber M, Rando OJ (January 2016). “Biogenesis and function of tRNA fragments during sperm maturation and fertilization in mammals”. Science 351 (6271): 391–396. Bibcode: 2016Sci...351..391S. doi:10.1126/science.aad6780. PMC 4888079. PMID 26721685.
- ^ Casas E, Cai G, Neill JD (2015). “Characterization of circulating transfer RNA-derived RNA fragments in cattle”. Frontiers in Genetics 6: 271. doi:10.3389/fgene.2015.00271. PMC 4547532. PMID 26379699.
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (July 2015). “Precise mapping and dynamics of tRNA-derived fragments (tRFs) in the development of Triops cancriformis (tadpole shrimp)”. BMC Genetics 16: 83. doi:10.1186/s12863-015-0245-5. PMC 4501094. PMID 26168920.
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (September 2015). “Age-driven modulation of tRNA-derived fragments in Drosophila and their potential targets”. Biology Direct 10: 51. doi:10.1186/s13062-015-0081-6. PMC 4572633. PMID 26374501.
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (August 2016). “MINTbase: a framework for the interactive exploration of mitochondrial and nuclear tRNA fragments”. Bioinformatics 32 (16): 2481–2489. doi:10.1093/bioinformatics/btw194. PMC 4978933. PMID 27153631.
- ^ Pliatsika V, Loher P, Magee R, Telonis AG, Londin E, Shigematsu M, Kirino Y, Rigoutsos I (January 2018). “MINTbase v2.0: a comprehensive database for tRNA-derived fragments that includes nuclear and mitochondrial fragments from all The Cancer Genome Atlas projects”. Nucleic Acids Research 46(D1) (D1): D152–D159. doi:10.1093/nar/gkx1075. PMC 5753276. PMID 29186503.
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (January 2015). “tRFdb: a database for transfer RNA fragments”. Nucleic Acids Research 43 (Database issue): D141-5. doi:10.1093/nar/gku1138. PMC 4383946. PMID 25392422.
- ^ a b Vincent RM, Wright BW, Jaschke PR (April 2019). “Measuring Amber Initiator tRNA Orthogonality in a Genomically Recoded Organism”. ACS Synthetic Biology 8 (4): 675–685. doi:10.1021/acssynbio.9b00021. PMID 30856316.
- ^ Govindan A, Miryala S, Mondal S, Varshney U (November 2018). “Development of Assay Systems for Amber Codon Decoding at the Steps of Initiation and Elongation in Mycobacteria”. Journal of Bacteriology 200 (22). doi:10.1128/jb.00372-18. PMC 6199473. PMID 30181124.
- ^ White RJ (March 1997). “Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control?”. Trends in Biochemical Sciences 22 (3): 77–80. doi:10.1016/S0968-0004(96)10067-0. PMID 9066256.
- ^ Sharp S, Dingermann T, Söll D (September 1982). “The minimum intragenic sequences required for promotion of eukaryotic tRNA gene transcription”. Nucleic Acids Research 10 (18): 5393–5406. doi:10.1093/nar/10.18.5393. PMC 320884. PMID 6924209.
- ^ a b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (December 2007). “The expanding RNA polymerase III transcriptome”. Trends in Genetics 23 (12): 614–622. doi:10.1016/j.tig.2007.09.001. hdl:11381/1706964. PMID 17977614.
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (December 2009). “Processing of multiple-intron-containing pretRNA”. Proceedings of the National Academy of Sciences of the United States of America 106 (48): 20246–20251. Bibcode: 2009PNAS..10620246T. doi:10.1073/pnas.0911658106. PMC 2787110. PMID 19910528.
- ^ Abelson J, Trotta CR, Li H (May 1998). “tRNA splicing”. The Journal of Biological Chemistry 273 (21): 12685–12688. doi:10.1074/jbc.273.21.12685. PMID 9582290.
- ^ a b Soma A (2014). “Circularly permuted tRNA genes: their expression and implications for their physiological relevance and development”. Frontiers in Genetics 5: 63. doi:10.3389/fgene.2014.00063. PMC 3978253. PMID 24744771.
- ^ Frank DN, Pace NR (1998). “Ribonuclease P: unity and diversity in a tRNA processing ribozyme”. Annual Review of Biochemistry 67 (1): 153–180. doi:10.1146/annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). “tRNase Z”. Protein and Peptide Letters 14 (2): 137–145. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Randau L, Schröder I, Söll D (May 2008). “Life without RNase P”. Nature 453 (7191): 120–123. Bibcode: 2008Natur.453..120R. doi:10.1038/nature06833. PMID 18451863.
- ^ Weiner AM (October 2004). “tRNA maturation: RNA polymerization without a nucleic acid template”. Current Biology 14 (20): R883-5. doi:10.1016/j.cub.2004.09.069. PMID 15498478.
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (February 1998). “Identification of a tRNA-specific nuclear export receptor”. Molecular Cell 1 (3): 359–369. doi:10.1016/S1097-2765(00)80036-2. PMID 9660920.
- ^ Arts GJ, Fornerod M, Mattaj IW (March 1998). “Identification of a nuclear export receptor for tRNA”. Current Biology 8 (6): 305–314. doi:10.1016/S0960-9822(98)70130-7. PMID 9512417.
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (December 1998). “The role of exportin-t in selective nuclear export of mature tRNAs”. The EMBO Journal 17 (24): 7430–7441. doi:10.1093/emboj/17.24.7430. PMC 1171087. PMID 9857198.
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (August 2003). “Possibility of cytoplasmic pre-tRNA splicing: the yeast tRNA splicing endonuclease mainly localizes on the mitochondria”. Molecular Biology of the Cell 14 (8): 3266–3279. doi:10.1091/mbc.E02-11-0757. PMC 181566. PMID 12925762.
- ^ Kühnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 March 2021). “tRNA sequences can assemble into a replicator”. eLife 10. doi:10.7554/eLife.63431. PMC 7924937. PMID 33648631.
- ^ Maximilian, Ludwig (3 April 2021). “Solving the Chicken-and-the-Egg Problem – "A Step Closer to the Reconstruction of the Origin of Life"”. SciTechDaily 3 April 2021閲覧。
- ^ Kresge, Nicole; Simoni, Robert D.; Hill, Robert L. (October 7, 2005). “The Discovery of tRNA by Paul C. Zamecnik”. Journal of Biological Chemistry 280 (40): e37–e39. doi:10.1016/S0021-9258(20)79029-0.
- ^ Clark BF (October 2006). “The crystal structure of tRNA”. Journal of Biosciences 31 (4): 453–457. doi:10.1007/BF02705184. PMID 17206065.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (March 1965). “Structure of a Ribonucleic Acid”. Science 147 (3664): 1462–1465. Bibcode: 1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761.
- ^ “Obituary”. The New York Times (July 4, 1991). 2022年9月25日閲覧。
- ^ “The Nobel Prize in Physiology or Medicine 1968: Robert W. Holley – Facts”. Nobel Prize Outreach AB (2022年). 18 March 2022閲覧。
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (November 1975). “Structure of yeast phenylalanine transfer RNA at 2.5 A resolution”. Proceedings of the National Academy of Sciences of the United States of America 72 (11): 4414–4418. Bibcode: 1975PNAS...72.4414L. doi:10.1073/pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (January 1973). “Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain”. Science 179 (4070): 285–288. Bibcode: 1973Sci...179..285K. doi:10.1126/science.179.4070.285. PMID 4566654.
外部リンク
[編集]- tRNAdb (updated and completely restructured version of Spritzls tRNA compilation)
- tRNA link to heart disease and stroke
- GtRNAdb: Collection of tRNAs identified from complete genomes
- HGNC: Gene nomenclature of human tRNAs
- Molecule of the Month © RCSB Protein Data Bank:
- Rfam entry for tRNA
- tRNADB-CE: tRNA gene database curated manually by experts
- PDBj入門≫今月の分子≫15 転移RNA
- PDBj入門≫今月の分子≫81 伸長因子


 French
French Deutsch
Deutsch