Virus ADN , la enciclopedia libre
Un virus ADN es un virus cuyo material genético está compuesto por ADN, no usando ARN como intermediario durante la replicación. Los virus que usan el ARN bien como material genético o como intermediario durante la replicación son virus ARN.[2] El ADN puede ser tanto de cadena simple (monocatenario) como de doble cadena (bicatenario), siendo estos últimos más diversos y frecuentes. La replicación dentro de las células depende de una ADN polimerasa dependiente del ADN (que lee el ADN). Es común que los ADN de cadena simple se expandan a ADN de cadena doble en las células infectadas.
Clasificación de Baltimore y replicación
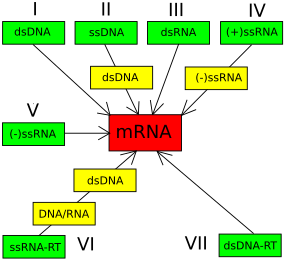
[editar]El sistema de clasificación de Baltimore se utiliza para agrupar virus en función de su forma de síntesis de ARN mensajero (ARNm) y, a menudo, se usa junto con la taxonomía de virus estándar, que se basa en la historia evolutiva. Los virus de ADN constituyen tres grupos de Baltimore: Grupo I: virus ADN bicatenario, Grupo II: virus ADN monocatenario y Grupo VII: virus ADN bicatenario retrotranscrito. Si bien la clasificación de Baltimore se basa principalmente en la transcripción del ARNm, los virus de cada grupo de Baltimore también suelen compartir su forma de replicación. Los virus en un grupo de Baltimore no necesariamente comparten una relación genética o morfológica.
Virus ADN bicatenario
[editar]El primer grupo de virus de ADN de Baltimore son los que tienen un genoma de ADN bicatenario. Todos los virus de ADN bicatenario tienen su ARNm sintetizado en un proceso de tres pasos. Primero, un complejo de preiniciación de la transcripción se une al ADN corriente arriba del sitio donde comienza la transcripción, lo que permite el reclutamiento de una ARN polimerasa del huésped. En segundo lugar, una vez que se recluta la ARN polimerasa, utiliza la hebra negativa como plantilla para sintetizar las hebras de ARNm. En tercer lugar, la ARN polimerasa termina la transcripción al alcanzar una señal específica, como un sitio de poliadenilación.
Los virus de ADN bicatenario utilizan varios mecanismos para replicar su genoma. La replicación bidireccional, que es la forma típica de replicación del ADN en eucariotas, se usa ampliamente. En la replicación bidireccional, se escinde un genoma circular para separar las dos hebras, creando una bifurcación desde la cual la replicación de ambas hebras progresa alrededor del genoma al mismo tiempo, yendo en dos direcciones opuestas hasta llegar al extremo opuesto. También se utiliza un mecanismo de círculo rodante que produce hebras lineales mientras avanza en un bucle alrededor del genoma circular que igualmente replica ambas hebras simultáneamente. En lugar de replicar ambas hebras a la vez, algunos virus de ADN bicatenario utilizan un método de desplazamiento de hebra mediante el cual una hebra se sintetiza a partir de una hebra molde y luego se sintetiza una hebra complementaria a partir de la hebra sintetizada anteriormente, formando un genoma de ADN bicatenario. Por último, algunos virus ADN bicatenario se replican como parte de un proceso llamado transposición replicativa mediante el cual un genoma viral en el ADN de una célula huésped se replica en otra parte de un genoma huésped.
Los virus de ADN bicatenario pueden subdividirse entre los que se replican en el núcleo y, como tales, son relativamente dependientes de la maquinaria de la célula huésped para la transcripción y replicación, y los que se replican en el citoplasma, en cuyo caso han evolucionado o adquirido sus propios medios para ejecutar la transcripción y la replicación.
Virus ADN monocatenario
[editar]El segundo grupo de virus de ADN de Baltimore son los que tienen un genoma de ADN monocatenario. Los virus de ADN monocatenario tienen la misma forma de transcripción que los virus de ADN bicatenario. Sin embargo, debido a que el genoma es monocatenario, primero una ADN polimerasa lo convierte en una forma bicatenaria al entrar en una célula huésped. Luego, el ARNm se sintetiza a partir de la forma bicatenaria. La forma bicatenaria de los virus de ADN monocatenario puede producirse directamente después de la entrada en una célula o como consecuencia de la replicación del genoma viral. Los virus de ADN monocatenario eucariotas se replican en el núcleo.
La mayoría de los virus de ADN monocatenario contienen genomas circulares que se replican mediante la replicación en círculo rodante (RCR). El ADN monocatenario RCR se inicia mediante una endonucleasa que se une a la hebra positiva y la escinde, lo que permite que una ADN polimerasa utilice la hebra negativa como molde para la replicación. La replicación progresa en un bucle alrededor del genoma mediante la extensión del extremo 3' de la hebra positiva, desplazando la hebra positiva anterior, y la endonucleasa escinde la hebra positiva de nuevo para crear un genoma independiente que se liga en un bucle circular. El nuevo ADN monocatenario puede empaquetarse en viriones o replicarse mediante una ADN polimerasa para formar una forma bicatenaria para la transcripción o continuación del ciclo de replicación.
Los parvovirus contienen genomas de ADN monocatenario lineales que se replican mediante la replicación en horquilla rodante (RHR), que es similar a RCR. Los genomas de parvovirus tienen bucles en forma de horquilla en cada extremo del genoma que se despliegan y repliegan repetidamente durante la replicación para cambiar la dirección de la síntesis de ADN y moverse hacia adelante y hacia atrás a lo largo del genoma, produciendo numerosas copias del genoma en un proceso continuo. Luego, los genomas individuales son escindidos de esta molécula por la endonucleasa viral. Para los parvovirus, la cadena de sentido positivo o negativo puede empaquetarse en cápsides, que varían de virus a virus.
Casi todos los virus de ADN monocatenario tienen genomas de sentido positivo, pero existen algunas excepciones y peculiaridades. La familia Anelloviridae es la única familia de ADN monocatenario cuyos miembros tienen genomas de sentido negativo, que son circulares. Los parvovirus, como se mencionó anteriormente, pueden empaquetar la cadena de sentido positivo o negativo en viriones.
Virus ADN bicatenario retrotranscrito
[editar]El séptimo grupo de Baltimore contiene virus que tienen un genoma de ADN de bicatenario que se replican por un intermedio de ARN en su ciclo de replicación. Los virus ADN bicatenario retrotranscrito tienen un espacio en una hebra, que se repara para crear un genoma de ADN bicatenario retrotranscrito completo antes de la transcripción. Los virus ADN bicatenario retrotranscrito se transcriben de la misma manera que los virus e ADN bicatenario, pero hacen uso de una transcripción inversa para replicar su genoma circular mientras aún está en la cápside. La ARN polimerasa II de la célula huésped transcribe las cadenas de ARN del genoma en el citoplasma y el genoma se replica a partir de estas cadenas de ARN. El genoma de ADN bicatenario retrotranscrito se produce a partir de hebras de ARN pregenómico mediante el mismo mecanismo general que los virus ARN monocatenario retrotranscrito, pero la replicación se produce en un bucle alrededor del genoma circular. Después de la replicación, el genoma de ADN bicatenario retrotranscrito puede empaquetarse o enviarse al núcleo para más rondas de transcripción.
Origen y evolución
[editar]Según el Comité Internacional de Taxonomía de Virus (ICTV) los virus de ADN se pueden encontrar en cinco de los seis dominios establecidos y pudieron tener diversos orígenes evolutivos.[3]
- Duplodnaviria: Contiene a los virus de ADN que tienen una proteína específica denominada HK97-MCP y una enzima única la terminasa dentro la cápside. Son virus de ADN bicatenario y con cápsides icosaedricas. Incluye los clásicos caudovirus de procariotas y sus descendientes eucariotas; los herpesvirus y mirusvirus. Se ha sugerido que surgieron antes que el último antepasado común universal (LUCA) y que los caudovirus precedieron a los herpesvirus y mirusvirus con la pérdida de la cola contráctil. La fusión de un replicón de ADN precelular con una ADN polimerasa, la terminasa, la HK97-MCP y proteínas de dominio DUF1884, hélice-hebra, dodecinas y encapsulinas llevó a la formación de los viriones de Duplodnaviria, que al principio no tendrían la cola contráctil, por lo que esta se habría desarrollado después de LUCA.
- Adnaviria: Incluye virus de arqueas que contienen una proteína específica dímera denominada SIRV2 dentro la cápside, morfología filamentosa y ADN en forma de A. Todos los representantes son virus de ADN bicatenario. Su origen es anterior al último antepasado común universal (LUCA) debido a la falta de homólogos celulares. La fusión de un replicón de ADN precelular con la ADN polimerasa, la SIRV2 y proteínas de dominio hélice-hélice, METT26, SER2, ILE26 y ARG128 llevó a la formación de los viriones de Adnaviria. Es también el único dominio que hasta ahora no tiene descendientes en los eucariotas.
- Varidnaviria: Contiene a los virus de ADN que tienen proteínas en rollo de gelatina vertical puede ser en doble rollo DJR-MCP o en rollo simple SJR-MCP dentro la cápside. Casi todos son de ADN bicatenario, salvo una familia que es de ADN monocatenario, pero que evolucionaron de virus de ADN bicatenario de este dominio. Los miembros de este dominio tienen mayormente cápsides icosaedricas, aunque en algunos miembros la cápside icosaedrica ha evolucionado a una forma ovoide o de ladrillo. Se divide en dos reinos: Bamfordvirae que incluye los virus con las proteínas en doble rollo de gelatina DJR-MCP entre los que se encuentran los adenovirus, los virus gigantes y varios virus procariotas como los tectivirus o turrivirus, mientras que Helvetiavirae incluye los virus con las proteínas en rollo simple de gelatina SJR-MCP que incluye virus procariotas que parecen haber dado origen a los virus de los Bamfordvirae mediante la unión de dos SJR-MCP en un solo pliegue originando la DJR-MCP. Se ha sugerido que el origen de Varidanviria también es anterior al del último antepasado común universal (LUCA) y que los eucariovirus del dominio descienden de los tectivirus a través de unos virus intermediaros denominados "polintovirus" que posteriormente se convertirían en los transposones polintones. La fusión de un replicón de ADN precelular con la ADN polimerasa y proteínas de dominio cupin, TNF, Pro-PD, CBM, nucleoplasminas y la SJR-MCP vertical llevó a la formación de los viriones de Varidnaviria. Los virus gigantes evolucionaron de virus pequeños descendientes de los polintovirus, que aumentaron su genoma y el tamaño del virión mediante la duplicación y deleción de genes, la inclusión de elementos genéticos móviles y la adquisición masiva de genes del huésped y sus bacterias endosimbióticas, incluidos los genes para la traducción y los genes informáticos que se consideran los más resistentes a la transferencia horizontal. Los virus gigantes pudieron haber dado origen a los virus inusuales de la clase Naldaviricetes o descender de un ancestro compartido.
- Monodnaviria: Contiene a los virus de ADN con una proteína específica denominada REP que hace una replicación en círculo rodante y una endonucleasa HUH. Incluye a casi todos los virus de ADN monocatenario junto con los papilomavirus y poliomavirus que son de ADN bicatenario, pero que evolucionaron de virus de ADN monocatenario. Los miembros de este dominio tienen cápsides icosaedricas, aunque algunos miembros tienen cápsides helicoidales. Se divide en cuatro reinos Shotokuvirae que incluye los eucariovirus del dominio como los papilomavirus, poliomavirus, circovirus, parvovirus, nanovirus, geminivirus, etc. Los demás reinos Sangervirae, Trapavirae y Loebvirae, están conformados exclusivamente por virus procariotas, Loebvirae se destaca por incluir los bacteriófagos filamentosos como los inovirus, mientras que los demás incluyen bacteriófagos icosaedricos sin cola como los microvirus. Se ha sugerido que los virus de este dominio surgieron a través de tres eventos en los que plásmidos se integraron en las cápsides de virus de ARN monocatenario positivo, remplazando el genoma y las enzimas típico del virus. Los eucariovirus del dominio (Shotokuvirae) descienden de bacteriófagos emparentados con los microvirus con los que están más estrechamente emparentados y comparten características estructurales. Los virus de Monodnaviria parecen haber originado la superfamilia de transposones helitrones y algunos tipos de plásmidos.
- Linaje de virus fusiformes y Clavaviridae: Se ha sugerido que las familias de virus fusiformes o en forma de limón de arqueas Fuselloviridae, Halspiviridae, Thaspiviridae, Itzamnaviridae y Bicaudaviridae están relacionadas por la presencia de una proteína homóloga la SSV1, ATPasas de la superfamilia AAA, forma de limón y el ensamblaje, la proteína homóloga también está presente en la familia de virus arqueanos filamentosos Clavaviridae, por lo que se ha sugerido que estos virus evolucionaron de virus filamentosos emparentados con Clavaviridae que alteraron su forma para almacenar un genoma más grande y conformarían un dominio o linaje. Su origen al igual que otros dominios es anterior al último antepasado común universal (LUCA).
- Riboviria: es un dominio que incluye todos los virus de ARN y virus retrotranscritos por lo que incluye a los virus de ADN bicatenario retrotranscritos a pesar de que todos los otros virus sean de ARN. Estos se caracterizan por tener las enzimas transcriptasa inversa y la ARN polimerasa dependiente de ARN (RdRP) que se cree que son las enzimas más primitivas que transcriben ácidos nucleicos. Los virus ADN bicatenario retrotranscritos que contiene dos familias los hepadnavirus y los caulimovirus que infectan animales y plantas se incluyen en el reino Pararnavirae junto con los virus de ARN monocatenario retrotranscritos. Se ha sugerido que estos dos grupos se originaron de un evento en el que un retrotransposón no LTR se integró en la cápside de un virus de ARN, remplazando el genoma y las enzimas típico del virus y que los virus ADN bicatenario retrotranscritos son polifiléticos siendo los caulimovirus una rama derivada de los virus ARN monocatenario retrotranscrito y los hepadnavirus la rama basal del reino. Los virus retrotranscritos pudieron surgir un poco después de que se originaran los eucariotas.
Además existen alrededor de 16 familias de virus de ADN no asignadas a ningún dominio, los cuales son virus de ADN bicatenario de arqueas con cápsides inusuales que son difíciles de clasificar debido a la falta de homología con otros virus de ADN, por tanto se considera que están desconectados de las virosfera. Se ha sugerido que los virus de ADN con cápsides inusuales de arqueas son descendientes de varios linajes extintos de virus de ADN que existieron antes que el último antepasado común universal (LUCA). Algunos virus inusuales de arqueas como Ampullaviridae, Guttaviridae, Bicaudaviridae, Fuselloviridae, Thaspiviridae y Halspiviridae parecen estar relacionados con plásmidos y transposones procariotas y de hecho sus genomas pueden haber evolucionado a partir de estos elementos genéticos móviles no virales. Las familias Ampullaviridae, Thaspiviridae y Halspiviridae muestran una relación estrecha con los transposones capsones, mientras que las familias Guttaviridae, Bicaudaviridae, y Fuselloviridae, están vinculadas con algunos tipos de plásmidos arqueales. Se ha planteado la posibilidad de que la enigmatica virosfera arqueal evoluciono mediante la recombinación genética entre elementos genéticos móviles no virales y virus. También queda sin asignar la familia de virus bacterianos de ADN inusuales Plasmaviridae que tampoco muestra relación filogenética con otros virus y organismos celulares, cuya procedencia podría ser similar a la que se propuso para los virus inusuales de arqueas.
Antiguamente se propuso que los virus de ADN junto a otros acelulares de ADN deberían reunirse bajo el grupo Deoxyviria, no obstante, aparentemente algunos virus de ADN están más estrechamente relacionados con los organismos celulares (Cytota) que a otros virus de ADN, lo cual vuelve parafilético.
Referencias
[editar]- ↑ Expression of animal virus genomes, by Baltimore D.
- ↑ a b N.J. Dimmock, A.J. Easton y K. Leppard, Introduction to Modern Virology, Ed. Wiley, 2006, ISBN 978-1-4051-3645-7.
- ↑ Eugene V. Koonin, Mart Krupovic, and Vadim I. Agol, 2021. The Baltimore Classification of Viruses 50 Years Later: How Does It Stand in the Light of Virus Evolution?. American Society of Microbiogy.


 French
French Deutsch
Deutsch